
|

|

|

|
関係データベースを演繹データベースに拡張することにより、遺伝病のデータを扱うことが可能になったが、分子生物学の多種多様なデータはこれで十分だろうか。分子生物学に限らず、様々なデータに対する複雑な操作を分かりやすく扱うために、オブジェクト指向データデータベース(object-oriented database)が注目されている。オブジェクト指向データモデルはケイ(A. Kay)の Smalltalk に代表されるオブジェクト指向プログラミングの考え方に基づくものである。
オブジェクト指向とは何かを説明するのに、「世界はオブジェクトの集まりである」という言葉がよく用いられる。何をオブジェクトにするかは実世界をどのようにモデル化するかに依存するが、オブジェクトの最も基本的な要件の1つは、データと手続きが一体化され、外部からのアクセスは定義されたプロトコルでしかできないようになっていることである。これをオブジェクトのカプセル化(encapsulation)という。オブジェクトに関するデータ操作はメソッド(method)と呼ばれ、外部からメッセージ(message)によりオブジェクトにメソッドの処理を依頼する。また、オブジェクトの識別性(object identity)という概念があり、オブジェクトを区別する識別子がまずあって、付随する性質が全く同じであっても識別子が異なれば異なるオブジェクトとなる。関係データベースでは属性の値がすべて同じであれば同じレコードとみなし、値に基づく表現がなされているが、オブジェクト指向データベースでは識別子に基づく表現がなされているのである。さらに、共通の性質や手続きをもったオブジェクトはクラス(class)として定義される。つまり、オブジェクトはテンプレートとしてのクラスとその実例であるインスタンス(instance)に分けることができる。クラスには階層を導入することができ、上位のクラスで定義された性質や手続きは下位のクラスへ自動的に継承(inheritance)される。
オブジェクト指向プログラミングではオブジェクト間のメッセージ通信によって計算をモデル化し、オブジェクト指向データベースではオブジェクト間の関係によってデータ構造をモデル化する。オブジェクト指向には多くの概念が含まれているが、関係データベースの上にオブジェクト指向的なユーザーインターフェースをつけることもできるわけで、ユーザーにとって関係データベースがよいかオブジェクト指向データベースがよいかといった議論はあまり意味がないかもしれない。ただしデータベースを作る立場からは、実世界の複雑なデータ構造を表現でき得る点で、オブジェクト指向の方が将来の可能性が高い。関係モデルが非正規関係モデル(入れ子関係モデル)へと発展したように、オブジェクトの延長に複合オブジェクト(complex object)を考えることができる。ただ現状では、複合オブジェクトに論理的な基礎があるわけではなく、実用的にリスト、タプル、セットなど、他のオブジェクトを属性としてもつ複合オブジェクトが定義されている。
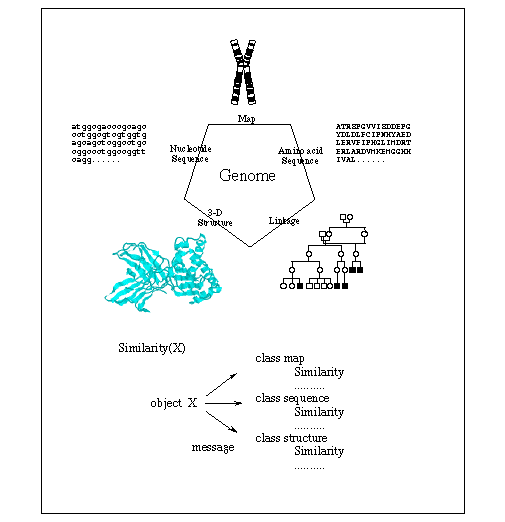
上図はオブジェクト指向ゲノムデータベースの概念図である。ゲノム関連データは染色体のイメージ、配列の文字、立体構造のグラフィックスなどマルチメディアであること、生物の系統樹や家系図、発生における遺伝子発現制御の上下関係を始め、複雑なデータ構造を扱う必要があることから、オブジェクト指向データベースにはうってつけの材料である。ここに示したように、ゲノムには染色体地図、塩基配列、アミノ酸配列、立体構造、リンケージといった側面があり、これらをいかに統一的に扱うかがデータベース化の問題である。例えば、ゲノムデータベースでは類似なものを検索することが頻繁に行われる。ゲノムの類似性には地図としての類似性、配列(塩基配列またはアミノ酸配列)としての類似性、立体構造の類似性があり、それぞれ異なる種類のデータに異なる計算手続きを適用する必要がある。ここでは類似性を探す操作はそれぞれのクラスでのメソッドとして定義されてカプセル化が行われ、ユーザーは類似性検索という同じ概念で、実際はデータの種類によって異なる検索が行われて、結果を得ることができる。