1991年度に発足した当時の文部省ヒト・ゲノムプログラムでは、生命の基本データであるゲノムを解読することにより、細胞、個体、生態系といった異なるレベルでの生命現象の理解と、医学、薬学、農学等の分野への応用を目指した研究が開始されました。そして、ゲノムを基盤とした新しい生命科学研究を推進するには、情報インフラストラクチャーの整備が不可欠であるとの認識から、京都大学化学研究所と東京大学医科学研究所を中心に、ゲノムネット(GenomeNet)と名づけたコンピュータネットワークの構築とデータベースサービスの運用が始まりました。インターネットの普及とともにネットワーク構築の必要性はなくなり、現在ではゲノムネットとは京都大学化学研究所バイオインフォマティクスセンターが提供するデータベースサービスを意味しています。 ゲノムネットでは、当初は我が国のインフラストラクチャー整備、すなわち既存の分子生物学データベースを統合的に利用できる環境を提供することに重点があり、そのためDBGET検索システムとそのWeb版を開発・運用してきました。分子生物学の分野では文献情報の他に、核酸の塩基配列、タンパク質のアミノ酸配列と立体構造、それに配列モチーフなど、すでに世界中で標準的なデータベースが作られていたからです。一方、ゲノムを基盤とした新しい生命科学研究には、高次の生命システムの機能情報に関するデータベースが必須であるとの認識から、1995年に京都大学化学研究所でKEGGと名づけた新しいデータベースプロジェクトが開始されました。KEGGはしだいにゲノムネットの中心システムとなり、同時にゲノムネットは海外からの利用者比率が8〜9割にも達する国際的なバイオ情報サービスとなっています。
ゲノムネットは以下のホームページアドレスから誰でも自由に利用することができます。
ゲノムネットが提供するサービスは大きく以下の3つに分けることができます。本書ではKEGGなど新しいゲノムネットサービスを中心に紹介しますが、従来型の分子生物学データベースの利用法も最初にとりあげます。
DBGETはゲノムネットのバックボーンとなる検索システムです。そこではデータベースはエントリーと呼ぶ単位の集合として取り扱われます。既存の分子生物学関連データベースのほとんどは、このような単純な見方で(すなわち、フラットファイルとして)眺めることができます。各エントリーにはエントリー名(またはアクセッション番号)というデータベース内でユニークな名前がつけられていますので、
 といった形の2項関係で表現されます。DBGET では LinkDB と呼ぶ2項関係だけのデータベースをもっており、2項関係を演繹して(組み合わせたり、逆向きにたどったりして)新たな2項関係を作ることにより、多くの関連データを容易に見いだせるようになっています。また、リンクはゲノムネットの外のデータベースにも多数つけられており、より専門的なデータベースで細かな情報を調べることができるようになっています。
といった形の2項関係で表現されます。DBGET では LinkDB と呼ぶ2項関係だけのデータベースをもっており、2項関係を演繹して(組み合わせたり、逆向きにたどったりして)新たな2項関係を作ることにより、多くの関連データを容易に見いだせるようになっています。また、リンクはゲノムネットの外のデータベースにも多数つけられており、より専門的なデータベースで細かな情報を調べることができるようになっています。
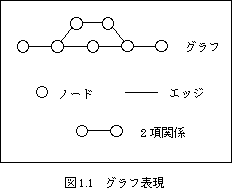
このようにDBGETシステムが取り扱うのは、多数のデータベースのエントリーの集合とそれらの間のリンク情報の集合です。これを分子生物学データのウェブと呼ぶことにしましょう。WWW (World Wide Web) がページをノードとし、ハイパーリンクをエッジとした膨大なグラフであるのと同様に、分子生物学データのウェブはエントリーをノード(頂点)とし、リンク情報をエッジ(辺)としたグラフとみなすことができます。ここでグラフとは数学的な概念で、図1.1に示したように、ノードとエッジの集合、または2項関係の集合のことです。次に述べるようにKEGGのデータオブジェクトはすべてグラフで表現されています(表1.1参照)。
表1.1 グラフで表現されたゲノムネットのデータオブジェクト
グラフ ノード エッジ WWW ページ ハイパーリンク DBGETにおける分子生物学データ
のウェブエントリー リンク情報 KEGGの遺伝子ユニバース 遺伝子 遺伝子間の関係(染色体上の隣接関係、
配列類似関係、共発現関係、他)KEGGのプロテインネットワーク タンパク質 一般化されたタンパク質間相互作用 KEGGのケミカルユニバース 化合物 化学反応
システマティックな配列決定により、2002年6月現在約90の生物種の全ゲノム配列が決定され、これらに含まれる遺伝子の数は約350万に達しています。KEGGの遺伝子ユニバースとは、この350万遺伝子をノードとし、それらの間の様々な関連を様々なタイプのエッジとした膨大なグラフのことです。遺伝子のウェブと言ってもいいでしょう。様々な関連とは、例えば配列類似性、オーソログ・パラログ等の進化的関連、マイクロアレイ発現プロフィール解析で得られる共発現の関係、酵母2ハイブリッドシステムその他の実験で得られるタンパク質(遺伝子産物)としての相互作用関係などです。このグラフの一部分(サブグラフ)が例えばヒトを構成する遺伝子群であり、あるいは生物種間で保存され共通のはたらきを司るオーソログ遺伝子群になるわけです。遺伝子ユニバースの情報は遺伝子カタログデータベースGENESや配列類似関係データベースSSDBなど表1.2に示したデータベースに蓄積されています。上に述べた通り、遺伝子ユニバースの各ノードは生物種名と遺伝子名(またはアクセッション番号)のペアで指定され、各エッジは
今後、全塩基配列が決定されたゲノムの数が増加するとともに、KEGGの遺伝子ユニバースに含まれるノードとエッジの数は飛躍的に増大していきます。しかしながら、生物種間で共通の遺伝子(オーソログ遺伝子)および共通の遺伝子ファミリー(パラログ遺伝子群のオーソログ関係)の数はそれほど増えないだろうとの期待があります。これらの遺伝子群をひとまとめにして新たなノード(サブグラフをノードにするのでコンパウンドノードあるいはネステッドノードともいいます)としたグラフを考えてみると、遺伝子ユニバースを低解像度で眺めたことになり、世界地図のような全体像が得られることになります。KEGGではこのような共通遺伝子群にKO (KEGG Orthology) とよぶアクセッション番号をつけています。生物種を越えた共通遺伝子群および共通遺伝子ファミリー群であるKOのユニバースが、生命現象を司る基本遺伝子セットになると考えられます。
表1.2.KEGGを構成するデータベース
グラフ データベース 内容 ソース 遺伝子
ユニバースGENES 全塩基配列が決定されたゲノムの遺伝子カタログ情報 GenBank、RefSeq等より自動変換後、KEGG独自のアノテーションを付加 GENOME 染色体地図に関する情報 SSDB 全塩基配列が決定されたすべての生物種のすべての遺伝子間の配列類似情報 GENESを用いてゲノム比較をSSEARCHで計算 EXPRESSION マイクロアレイによる遺伝子発現情報 我が国の研究コミュニティからの登録データ BRITE 遺伝子またはタンパク質の2項関係データ 酵母2ハイブリッドシステムなどで、文献等に公表されたデータ プロテイン
ネットワークPATHWAY タンパク質間相互作用ネットワークとして表現された細胞機能の知識 文献等から手作業で入力 ケミカル
ユニバースLIGAND 化学物質と化学反応に関する情報 文献等から手作業で入力
一方、KEGGのプロテインネットワークとは、細胞レベルでの生命現象を司る分子間相互作用ネットワークを、その主役であるタンパク質(厳密にはRNAを含むので遺伝子産物)のネットワークとして抽象化したものです。具体的には、代謝系や様々な制御系に関与するパスウェイ情報と、細胞の様々なプロセッシングに関与する分子複合体(コンプレックス)情報が、タンパク質をノードとし、タンパク質間の直接的および間接的な相互作用をエッジとしたグラフとして表現されています。つまり、これまで分子・細胞生物学、生化学、遺伝学などの実験で得られた広範な知識を、計算可能な形に表現したのがKEGGのプロテインネットワークです。このコンピュータ化の作業はすべて人手で行い、PATHWAYデータベースとして集大成されています。プロテインネットワークは低解像度から高解像度まで3段階の地図に相当する階層構造をしており、トップレベルは代謝、遺伝情報処理、環境情報処理、細胞プロセス、ヒトの病気の5つに分類されています。
プロテインネットワークも究極的には膨大なグラフになるはずですが、現時点での我々の知識は非常に限られています。KEGGの最大の目的は、ゲノム解析やプロテオーム解析から得られるデータ、すなわち遺伝子ユニバース(あるいはKOのユニバース)から新たなパスウェイやコンプレックスを予測して、既知のプロテインネットワークを広げていくことです。
ゲノムやプロテオームの情報を解読する際、もう1つの重要な手掛かりが、環境情報としての化学情報です。KEGGでは生体内の化学反応系および生体と相互作用する化学反応系を表現するために、生命現象に関与するあらゆる化合物をノードとし、それらの間の反応性をエッジとしたグラフを考え、これをケミカルユニバースと呼んでいます。ケミカルユニバースの情報は、これも現時点では非常に限られたものですが、LIGANDデータベースに蓄積されています。
代謝系のプロテインネットワークは酵素をノードとし、連続した化学反応を触媒する2つの酵素間にエッジがあるとみなしています。つまり最初の反応の生成物で同時に次の反応の基質である化合物が、エッジの情報になっています。ケミカルユニバースは逆に化合物がノードで反応(酵素)がエッジです。すなわち、プロテインネットワークとケミカルユニバースはノードとエッジの関係を入れ替えたグラフになっているわけで、この観点から両者を統合的に解析することができます。
ゲノムネットが提供している主なデータベースと各種サービスの一覧(2002年12月現在)は以下の通りです。
| データベース | 内容 | 作成者 | ||
| * | DNA | (GenBank+EMBLの複合データベース) | ||
| * | Protein | (SwissProt+PIR+PRF+PDBSTRの複合データベース) | ||
| * | nr-nt | 重複を除いた塩基配列 (GenBank, EMBL から作成) | ||
| * | nr-aa | 重複を除いたアミノ酸配列 (SwissProt, PIR, PRF, GenPeptから作成) | ||
| * | RefSeq | 核酸塩基配列 | 米国NCBIのRefSeqプロジェクト | |
| * | GenBank | 核酸塩基配列(DDBJを含む) | 米国NCBI、国立遺伝学研究所 | |
| * | EMBL | 核酸塩基配列 | 欧州EBI | |
| * | SwissProt | タンパク質アミノ酸配列 | スイスSIB、欧州EBI | |
| PIR | タンパク質アミノ酸配列 | 米国NBRF | ||
| PRF | タンパク質アミノ酸配列 | 蛋白質研究奨励会 | ||
| * | PDB | タンパク質等立体構造 | 米国RCSB | |
| * | PDBSTR | PDBアミノ酸配列 (PDBから作成) | ||
| EPD | 真核生物プロモーター | スイスISREC | ||
| TRANSFAC | 転写因子 | ドイツGBF | ||
| MotifDic | (PROSITE+BLOCKS+PRINTS+ProDom+Pfamの複合データベース) | |||
| PROSITE | タンパク質配列モチーフ | スイスSIB | ||
| BLOCKS | タンパク質配列モチーフ | Fred Hutchinson Cander Res. Ctr. | ||
| PRINTS | タンパク質配列モチーフ | University College London | ||
| ProDom | タンパク質配列モチーフ | フランスINRA | ||
| Pfam | タンパク質配列モチーフ | Wash.U.St.Louis & Sanger Ctr. | ||
| * | LIGAND | (COMPOUND+REACTION+ENZYMEの複合データベース) | ||
| * | COMPOUND | 生体に関与する化合物 | 京都大学化学研究所 | |
| * | REACTION | 生体に関与する化学反応 | 京都大学化学研究所 | |
| ENZYME | 酵素反応とその分類 | IUBMB, 京都大学化学研究所 | ||
| * | PATHWAY | KEGGパスウェイマップ | 京都大学化学研究所 | |
| * | GENES | KEGG遺伝子カタログ | 京都大学化学研究所 | |
| GENOME | KEGGゲノムカタログ | 京都大学化学研究所 | ||
| * | SSDB | GENESの配列類似関係 | 京都大学化学研究所 | |
| EXPRESSION | マイクロアレイによる遺伝子発現プロフィール | 京都大学化学研究所 | ||
| BRITE | 分子間相互作用 | 京都大学化学研究所 | ||
| * | OMIM | 遺伝病 | ジョンズホプキンス大学, NCBI | |
| PMD | 変異タンパク質 | 国立遺伝学研究所 | ||
| AAindex | アミノ酸指標 | 京都大学化学研究所 | ||
| LITDB | タンパク質関連文献 | 蛋白質研究奨励会 | ||
| * | Medline | 医学・生物学文献(リンクのみ) | NCBI, NLM | |
| * | LinkDB | リンク情報 | 京都大学化学研究所 | |
上記のデータベースのうち、nr-ntとnr-aaはホモロジー検索でのみ利用できます。またSSDBとBRITEには専用のデータベースシステムがあります。これら以外はすべてDBGETで検索が可能です。また、KEGGを構成するデータベース(LIGANDからBRITEまで)はDBGET以外にKEGGシステムとして統合されています。COMPOUNDとREACTIONはISISデータベースとして管理されており、Chemscapeを通して部分構造検索などの利用もできます。
| システム | 内容 | アドレス | 作成者 | |
| DBGET | データベース統合検索 | www.genome.ad.jp/dbget/ | 京大化研 | 2章 |
| STAG | DBGET全文検索 | stag.genome.ad.jp | 北陸先端大 | 2章 |
| BLAST | ホモロジー検索 | blast.genome.ad.jp | NCBI | 3章 |
| FASTA | ホモロジー検索 | fasta.genome.ad.jp | W.Pearson | 3章 |
| MOTIF | タンパク質モチーフ検索 | motif.genome.ad.jp | 京大化研 | 4章 |
| CLUSTALW | マルチプルアライメント | clustalw.genome.ad.jp | D.Higgins | 5章 |
| KEGG | パスウェイとゲノムの統合解析 | www.genome.ad.jp/kegg/ | 京大化研 | 6,7章 |
| SSDB | オーソログ、パラログの検索 | ssdb.genome.ad.jp | 京大化研 | 8章 |
| LIGAND | 化合物と化学反応の検索 | www.genome.ad.jp/ligand/ | 京大化研 | 9章 |
| BSORF | 枯草菌ゲノムデータベース | bacillus.genome.ad.jp | 奈良先端大 | 10章 |
| CYORF | らん藻ゲノムデータベース | cyano.genome.ad.jp | らん藻研究者 | 10章 |
| EXPRESSION | 発現プロフィール解析 | www.genome.ad.jp/kegg/expression/ | 京大化研 | 11章 |
| BRITE | 二項関係と推論 | www.genome.ad.jp/brite/ | 京大化研 | |