DBGET とは
DBGET は金久研究室が開発したデータベース検索システムで、ゲノムネットのバックボーンシステムとして利用されています(開発の歴史は英語版をご覧ください)。そこではデータベースはエントリーという単位の集合として取り扱われます。既存の分子生物学関連データベースのほとんどは、このような単純な見方で(すなわち、フラットファイルとして)眺めることができます。各エントリーにはエントリー名(またはアクセッション番号)というデータベース内でユニークな名前がつけられていますので、データベース名:エントリー名の組を与えると、世界中に存在する数多くのデータベースを統合的に参照することができるわけです。また、KEGG にある生物種ごとの遺伝子やタンパク質は
生物種名:遺伝子名により、やはり DBGET で統合的に検索することができます。
分子生物学の分野では異なるデータベースに関連するデータがあれば、それにリンクを付加してデータベース化が行われています。文献データと文献に報告された配列データとの関連、塩基配列とそれを翻訳したアミノ酸配列の関連などが代表例です。このリンク情報は
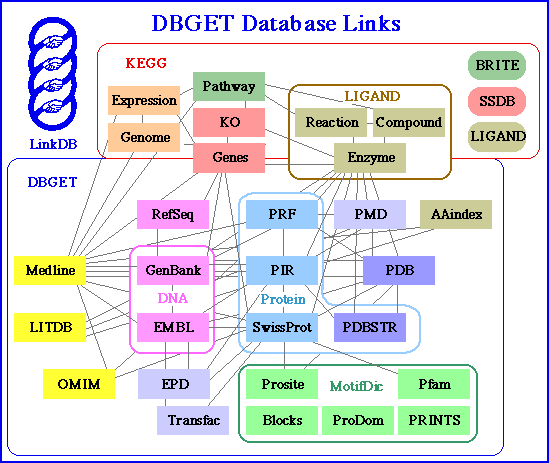
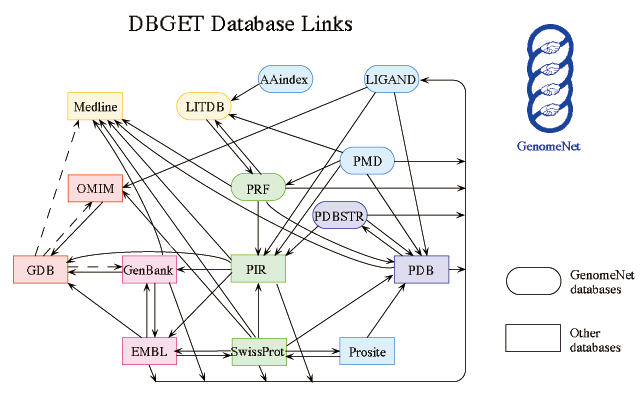
データベース1:エントリー1 → データベース2:エントリー2の形で表現され、これを2項関係といいます。DBGET では LinkDB と呼ぶ2項関係だけのデータベースをもっており、これにはゲノムネット内部だけでなく外部データベースへのリンク(主に KEGG からの)も含まれています。下図にある通り、当初 15 個程度のデータベースで始まったゲノムネットの DBGET では、現在は外部データベースを入れると数百ものデータベースが統合されています。
15 databases in September 1994
32 databases in September 2004