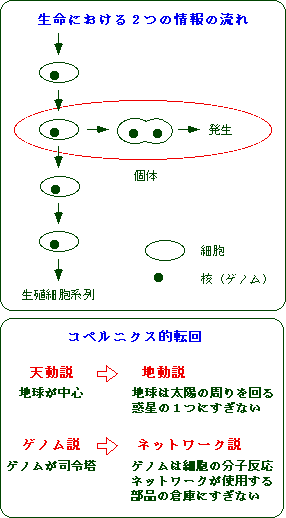
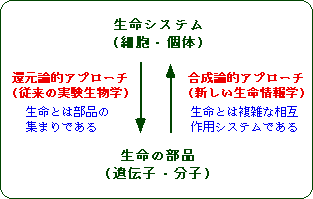
 20世紀後半の生命科学は分子生物学全盛の時代でした。その根底には「生命のはたらきは分子や遺伝子のはたらきとして理解できる」とする還元論の考え方があります。その究極の形として1980年代の終わりに始まったヒトゲノム計画では、ヒトをはじめ数多くの生物種において、ゲノムを解読する研究が行われています。ただし解読といっても、これは単にゲノムの全塩基配列を決定することであり、個々の遺伝子がどこにあるか、さらにそのはたらきが何であるかが、直ちに明らかになるわけではありません。実際、これまで全ゲノムが決定された比較的単純な生物種でも、ゲノム中に含まれる遺伝子のうち機能が分かっているものは半分以下しかありません。
20世紀後半の生命科学は分子生物学全盛の時代でした。その根底には「生命のはたらきは分子や遺伝子のはたらきとして理解できる」とする還元論の考え方があります。その究極の形として1980年代の終わりに始まったヒトゲノム計画では、ヒトをはじめ数多くの生物種において、ゲノムを解読する研究が行われています。ただし解読といっても、これは単にゲノムの全塩基配列を決定することであり、個々の遺伝子がどこにあるか、さらにそのはたらきが何であるかが、直ちに明らかになるわけではありません。実際、これまで全ゲノムが決定された比較的単純な生物種でも、ゲノム中に含まれる遺伝子のうち機能が分かっているものは半分以下しかありません。
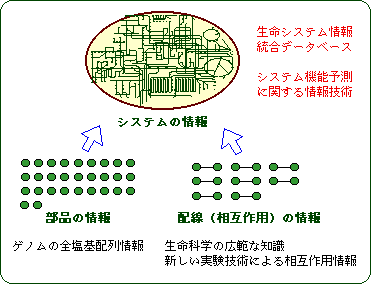 そもそもバイオインフォマティクスとは、ゲノム解析に伴う大量データ処理のために必然的に生まれたもので、当初は実験プロジェクトをサポートすることが主目的でした。一方、新しいポストゲノム時代のバイオインフォマティクスでは、ゲノムの情報を基盤に生命の原理を明らかにし、新産業の創出を行うことが目的で、情報科学が主役となります。ただし、これは新しい実験科学と融合した情報科学でなければなりません。ゲノムの配列情報とは基本的に部品(遺伝子あるいは分子)のカタログ情報であり、DNAチップやプロテオーム解析等の新しい実験技術に基づく部品間のつながり(相互作用)の情報がなければ、生命システムの再構築はできないからです。同時にこれまでに蓄積された膨大な生命科学の知識を相互作用情報という観点から体系化し、コンピュータ化することが極めて重要です。
そもそもバイオインフォマティクスとは、ゲノム解析に伴う大量データ処理のために必然的に生まれたもので、当初は実験プロジェクトをサポートすることが主目的でした。一方、新しいポストゲノム時代のバイオインフォマティクスでは、ゲノムの情報を基盤に生命の原理を明らかにし、新産業の創出を行うことが目的で、情報科学が主役となります。ただし、これは新しい実験科学と融合した情報科学でなければなりません。ゲノムの配列情報とは基本的に部品(遺伝子あるいは分子)のカタログ情報であり、DNAチップやプロテオーム解析等の新しい実験技術に基づく部品間のつながり(相互作用)の情報がなければ、生命システムの再構築はできないからです。同時にこれまでに蓄積された膨大な生命科学の知識を相互作用情報という観点から体系化し、コンピュータ化することが極めて重要です。
| 区分 | データベース | データ解析 | アルゴリズム |
|---|---|---|---|
| 分子 | 分子の構造データベース
・塩基配列(GenBank, EMBL, DDBJ) ・アミノ酸配列(SwissProt, PIR, PRF) ・立体構造(PDB) 分子の機能データベース ・核酸モチーフ(EPD, Transfac) ・タンパク質モチーフ(Prosite, Pfam) ・遺伝子アノテーション(KEGG, GO) | 配列/立体構造解析
・配列比較 ・立体構造比較 ・立体構造予測 機能部位解析 ・モチーフ抽出 ・モチーフ検索 ・機能予測 | 最適化アルゴリズム
・ダイナミックプログラミング(DP) ・シミュレーテッドアニーリング(SA) ・遺伝的アルゴリズム(GA) パターン認識・学習アルゴリズム ・ニューラルネットワーク(ANN) ・隠れマルコフモデル(HMM) ・サポートベクターマシン(SVM) |
| ゲノム (分子の集合) | ゲノムの機能データベース
・オーソロググループ(KEGG, COG) ・発現プロフィール ・遺伝子多型 | 比較ゲノム解析
トランスクリプトーム解析 プロテオーム解析 多型情報解析 | クラスタリングアルゴリズム
・階層的クラスター解析 ・コホーネンネットワーク |
| 相互作用 | 分子間相互作用データベース
・タンパク質間相互作用 ・二項関係(BRITE) 化学情報データベース ・化合物/化学反応(LIGAND) | ネットワーク解析
・パス計算 ・ネットワーク比較 ・ネットワーク予測 ・細胞シミュレーション ・パスウェイ工学 ・演繹データベース | グラフ比較アルゴリズム
・同型グラフ(クリーク) ・相関クラスター グラフ特徴抽出アルゴリズム ・準完全サブグラフ ・ハブ、オーソリティ グラフ計算アルゴリズム |
| ネットワーク (相互作用の集合) | パスウェイデータベース
・代謝系/制御系(KEGG) |
| システム | 内容 | ノード | エッジ |
|---|---|---|---|
| タンパク質 | 原子のネットワーク | 原子 | 原子間相互作用 |
| 細胞 | 分子のネットワーク | 分子 | 分子間相互作用 |
| 脳・神経系 | 細胞のネットワーク | 細胞 | 細胞間相互作用 |
| 生態系 | 個体のネットワーク | 個体 | 個体間相互作用 |
| 文化 | 人のネットワーク | 人 | 人間相互作用 |
物理学は自然界を異なるレベルで記述し、その原理を明らかにしてきました。バイオインフォマティクスは生物界を異なるレベルで記述し、生命の情報構築原理を明らかにしていくことでしょう。生命が地球上で誕生し進化してきたことを考えると、バイオインフォマティクスの究極の目標は、生物界と自然界をつなぐ大統一理論の構築と言えるかもしれません。
| 機 関 | アドレス | 主要検索システム | 主要データベース |
|---|---|---|---|
| ゲノムネット(京都大学化学研究所) | www.genome.ad.jp | DBGET | KEGG |
| NCBI(米国バイオテクノロジー情報センター) | www.ncbi.nlm.nih.gov | Entrez, BLAST | PubMed, GenBank |
| EBI(欧州バイオインフォマティクス研究所) | www.ebi.ac.uk | SRS | EMBL, SWISS-PROT |
| SIB(スイスバイオインフォマティクス研究所) | www.expasy.ch | SRS | SWISS-PROT |
ところで、大量かつ多様なバイオ情報は、上にあるような世界中のサーバーからインターネットを通じて誰でも自由に入手することができます。ここでも、米国バイオテクノロジー情報センター(NCBI)が国際的に圧倒的な力をもっていますが、現状ではこれは文献情報と配列情報だけです。京都大学化学研究所では、機能情報を制するものがバイオ情報全体を制することを予期し、また機能とは部品に還元できるものではなくシステムの属性として表現するべきものとの観点から、生命システム情報統合データベース KEGG を構築し、ゲノムネットの中心システムとして提供してきました。世界的にもまだ未発達の生命システム情報の基盤データベースを構築し、それを公共化することは、我が国にとって知的所有権を確保することであり、ゲノムから有用性を見いだす情報技術力で優位に立つことができ、経済の発展と社会の福祉に貢献できると考えています。

参考文献
M. Kanehisa, "Post-genome Informatics", Oxford University Press (2000)